|
Даю задания для студентов. Они хотят повторно пройти курс. Как отписаться от выполненного курса, что бы пройти его заново? |
Визуализация научных исследований
Пример 1. Поведение частиц в гравитационных колодцах (sample_1)
На сайте MicrosoftInternetExplorerTestDrive (http://ie.microsoft.com/testdrive/Graphics/TouchEffects/) представлен очень интересный пример, визуализирующий поведение частиц в гравитационных колодцах и действие сил магнитного отталкивания.
Запустите пример и испытайте его в разных режимах.
Пример 2. Моделирование поведения частиц жидкости (sample_2)
На сайте http://spielzeugz.de/html5/liquid-particles.html представлен пример, демонстрирующий поведение частиц жидкости.
Запустите пример. Обратите внимание на то, как можно управлять поведением частиц, двигая курсор мыши.
Пример 3. Поведение частиц в магнитном поле (sample_3)
На сайте норвежской студии дизайна Norges (http://www.norgeskreativefagskole.no/) представлены несколько примеров, визуализирующих различные физические процессы. В частности, в предлагаемом примере (sample_3) показано движение частиц вблизи источников магнитного поля.
Запустите пример. Посмотрите, как меняется поведение частиц при перемещении источников магнитного поля. Попробуйте сами добавить новые источники магнитного поля.
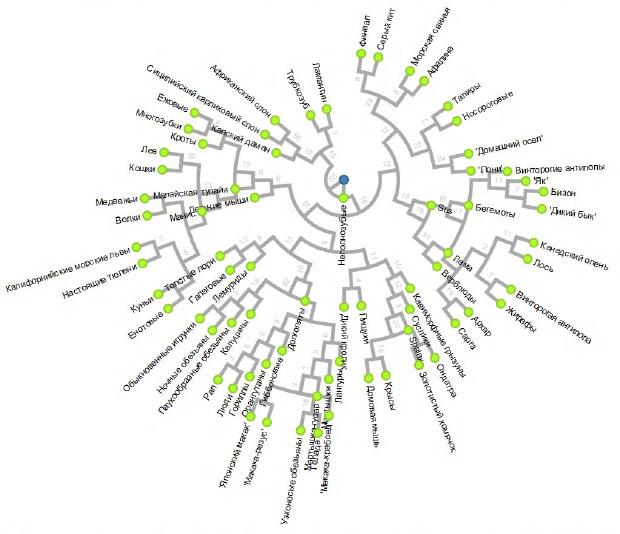
Пример 4. Филогенетическое дерево (sample_4)
Филограмма представляет собой диаграмму в виде разветвленного дерева, которая обобщает в графическом виде эволюционную историю группы организмов. При этом длина каждой ветви демонстрирует величину эволюции, обычно измеренную в количестве нуклеотидных остатков, наблюдаемых между точками соединенных вместе ветвей.
В 1986 году Майкл Миямото и Моррис Гудман опубликовали филограмму, показывающую результаты их работы по оценке исторических эволюционных связей в широкой выборке плацентарных млекопитающих.
Горизонтальные линии в дереве показывают степень эволюции вдоль ветви. Промежутки, вертикальные линии, хранят только информацию об общем предке. Длина промежутка не несет информации.
Филограмма может быть представлена в виде текстовой строки для обмена данными между программами и базами данных.
В нашем случае используется строка формата Newick (http://en.wikipedia.org/wiki/Newick_format):
(Неполнозубые:55, (((Трубкозуб:12, Ламантин:43):1, (Капский даман:29, (Сицилийский карликовый слон:18,
Африканский слон:5):55):10):15, (((Летучие мыши:27,
(Малайская тупайя:43, ((Кроты:24, (Многозубки:24, Ежовые:58):6):4, (Манис:5, ((Кошки:13, Лев:7):32, ((Волки:37,
Медвежьи:12):4, ((Настоящие тюлени:19,
Калифорнийские морские львы:17):7, (Енотовые:12, Куньи:22):9):5):17):13):3):10):6):8, (((Лемуриды:46,
(Галаговые:16, Толстые лори:27):10):8, (Долгопяты:15,
((Капуцины:10, (Паукообразные обезьяны:5, (Ночные обезьяны:6, Обыкновенные игрунки:9):3):3):18,
((Гиббоновые:6, (Орангутаны:1, (Гориллы:2,
(Люди:22, Pan:1):2):6):2):5, (Лангуры:1, (Мартышки:1, (Мартышка-гусар:0, (Макака-крабоед:2,
(Макака-резус:5, Японский макак:0):1, (Гелада:2,
Узконосые обезьяны:4):15):2):1):8):6):9):22):10):13, ((Пищухи:7, Дикий кролик:54):4,
(Кавиморфные грызуны:107, (Суслики:29, (Spalax:23, ((Крысы:71,
Домовая мышь:19):15, (Ондатра:27, Золотистый хомячок:32):27):20):8):12):15):11):9):12,
((Sus:50, ((Лама:10, Верблюды:24):31, (Бегемоты:31, (((Архар:9,
Capra:8):19, ((Винторогая антилопа:13, Жирафы:14):7, (Канадский олень:8, Лось:9):11):2):9,
(Винторогие антилопы:6, (Як:6, (Бизон:5,
Дикий бык:15):7):11):6):47):10):19):18, (((Домашний осел:6, Пони:31):24, (Тапиры:19, Носороговые:23):11):22,
((Морская свинья:4, Афалина:17):5, (Финвал:36,
Серый кит:2):8):29):12):12):16):55);
Задание:
Разработайте филогенетическое дерево жизни на основе следующей строки:
(((((((((((((((((((Escherichia_coli_EDL933:0.00000,Escherichia_coli_O157_H7:0.00000)96:0.00044,
((Escherichia_coli_O6:0.00000,Escherichia_coli_K12:0.00022)
76:0.00022,(Shigella_flexneri_2a_2457T:0.00000,Shigella_flexneri_2a_301:0.00000)100:0.00266)
75:0.00000)100:0.00813,((Salmonella_enterica:0.00000,
Salmonella_typhi:0.00000)100:0.00146,Salmonella_typhimurium:0.00075)100:0.00702)100:0.03131,
((Yersinia_pestis_Medievalis:0.00000,(Yersinia_pestis_KIM:0.00000,
Yersinia_pestis_CO92:0.00000)31:0.00000)100:0.03398,Photorhabdus_luminescens:0.05076)
61:0.01182)98:0.02183,((Blochmannia_floridanus:0.32481,
Wigglesworthia_brevipalpis:0.35452)100:0.08332,(Buchnera_aphidicola_Bp:0.27492,
(Buchnera_aphidicola_APS:0.09535,Buchnera_aphidicola_Sg:0.10235)100:0.10140)
100:0.06497)100:0.15030)100:0.02808,((Pasteurella_multocida:0.03441,Haemophilus_influenzae:0.03754)
94:0.01571,Haemophilus_ducreyi:0.05333)100:0.07365)100:0.03759,
((((Vibrio_vulnificus_YJ016:0.00021,Vibrio_vulnificus_CMCP6:0.00291)100:0.01212,
Vibrio_parahaemolyticus:0.01985)100:0.01536,Vibrio_cholerae:0.02995)
100:0.02661,Photobacterium_profundum:0.06131)100:0.05597)81:0.03492,Shewanella_oneidensis:0.10577)
100:0.12234,((Pseudomonas_putida:0.02741,Pseudomonas_syringae:
0.03162)100:0.02904,Pseudomonas_aeruginosa:0.03202)100:0.14456)98:0.04492,
((Xylella_fastidiosa_700964:0.01324,Xylella_fastidiosa_9a5c:0.00802)100:0.10192,
(Xanthomonas_axonopodis:0.01069,Xanthomonas_campestris:0.00934)100:0.05037)100:0.24151)
49:0.02475,Coxiella_burnetii:0.33185)54:0.03328,((((Neisseria_meningitidis_A:
0.00400,Neisseria_meningitidis_B:0.00134)100:0.12615,Chromobacterium_violaceum:0.09623)
100:0.07131,((Bordetella_pertussis:0.00127,(Bordetella_parapertussis:
0.00199,Bordetella_bronchiseptica:0.00022)67:0.00006)100:0.14218,Ralstonia_solanacearum:0.11464)100:0.08478)
75:0.03840,Nitrosomonas_europaea:0.22059)100:0.08761)
100:0.16913,((((((Agrobacterium_tumefaciens_Cereon:0.00000,Agrobacterium_tumefaciens_WashU:0.00000)
100:0.05735,Rhizobium_meliloti:0.05114)100:0.05575,
((Brucella_suis:0.00102,Brucella_melitensis:0.00184)100:0.08660,Rhizobium_loti:0.09308)51:0.02384)
100:0.08637,(Rhodopseudomonas_palustris:0.04182,
Bradyrhizobium_japonicum:0.06346)100:0.14122)100:0.05767,Caulobacter_crescentus:0.23943)
100:0.11257,(Wolbachia_sp._wMel:0.51596,(Rickettsia_prowazekii:0.04245,
Rickettsia_conorii:0.02487)100:0.38019)100:0.12058)100:0.12365)100:0.06301,((((Helicobacter_pylori_J99:0.00897,
Helicobacter_pylori_26695:0.00637)100:0.19055,
Helicobacter_hepaticus:0.12643)100:0.05330,Wolinella_succinogenes:0.11644)100:0.09105,
Campylobacter_jejuni:0.20399)100:0.41390)82:0.04428,
((Desulfovibrio_vulgaris:0.38320,(Geobacter_sulfurreducens:0.22491,Bdellovibrio_bacteriovorus:0.45934)
43:0.04870)69:0.04100,(Acidobacterium_capsulatum:0.24572,
Solibacter_usitatus:0.29086)100:0.20514)64:0.04214)98:0.05551,((Fusobacterium_nucleatum:0.45615,
(Aquifex_aeolicus:0.40986,Thermotoga_maritima:0.34182)
100:0.07696)35:0.03606,(((Thermus_thermophilus:0.26583,Deinococcus_radiodurans:0.29763)
100:0.24776,Dehalococcoides_ethenogenes:0.53988)35:0.04370,
((((Nostoc_sp._PCC_7120:0.12014,Synechocystis_sp._PCC6803:0.15652)98:0.04331,
Synechococcus_elongatus:0.13147)100:0.05040,(((Synechococcus_sp._WH8102:0.06780,
Prochlorococcus_marinus_MIT9313:0.05434)100:0.04879,Prochlorococcus_marinus_SS120:0.10211)
74:0.04238,Prochlorococcus_marinus_CCMP1378:0.16170)100:0.20442)
100:0.07646,Gloeobacter_violaceus:0.23764)100:0.24501)39:0.04332)51:0.02720)74:0.03471,
((((Gemmata_obscuriglobus:0.36751,Rhodopirellula_baltica:0.38017)
100:0.24062,((Leptospira_interrogans_L1-130:0.00000,Leptospira_interrogans_56601:0.00027)100:0.47573,
((Treponema_pallidum:0.25544,Treponema_denticola:0.16072)
100:0.19057,Borrelia_burgdorferi:0.42323)100:0.20278)95:0.07248)42:0.04615,
(((Tropheryma_whipplei_TW08/27:0.00009,Tropheryma_whipplei_Twist:0.00081)
100:0.44723,Bifidobacterium_longum:0.29283)100:0.14429,(((((Corynebacterium_glutamicum_13032:0.00022,
Corynebacterium_glutamicum:0.00000)100:0.03415,
Corynebacterium_efficiens:0.02559)100:0.03682,Corynebacterium_diphtheriae:0.06479)100:0.13907,
(((Mycobacterium_bovis:0.00067,
(Mycobacterium_tuberculosis_CDC1551:0.00000,Mycobacterium_tuberculosis_H37Rv:0.00000)98:0.00022)
100:0.03027,Mycobacterium_leprae:0.05135)97:0.01514,
Mycobacterium_paratuberculosis:0.02091)100:0.11523)100:0.09883,(Streptomyces_avermitilis:0.02680,
Streptomyces_coelicolor:0.02678)100:0.16707)91:0.06110)
100:0.26800)23:0.03480,((Fibrobacter_succinogenes:0.51984,(Chlorobium_tepidum:0.37204,
(Porphyromonas_gingivalis:0.11304,Bacteroides_thetaiotaomicron:0.13145)
100:0.34694)100:0.09237)62:0.04841,(((Chlamydophila_pneumoniae_TW183:0.00000,
(Chlamydia_pneumoniae_J138:0.00000,(Chlamydia_pneumoniae_CWL029:0.00000,
Chlamydia_pneumoniae_AR39:0.00000)37:0.00000)44:0.00000)100:0.10482,Chlamydophila_caviae:0.05903)
98:0.04170,(Chlamydia_muridarum:0.01938,
Chlamydia_trachomatis:0.02643)100:0.06809)100:0.60169)32:0.04443)67:0.04284)66:0.02646,
((Thermoanaerobacter_tengcongensis:0.17512,
((Clostridium_tetani:0.10918,Clostridium_perfringens:0.11535)78:0.03238,
Clostridium_acetobutylicum:0.11396)100:0.15056)100:0.11788,
(((((Mycoplasma_mobile:0.27702,Mycoplasma_pulmonis:0.28761)100:0.28466,
((((Mycoplasma_pneumoniae:0.10966,Mycoplasma_genitalium:0.11268)
100:0.31768,Mycoplasma_gallisepticum:0.24373)100:0.14180,Mycoplasma_penetrans:0.34890)
94:0.06674,Ureaplasma_parvum:0.33874)100:0.19177)
100:0.07341,Mycoplasma_mycoides:0.37680)100:0.12541,Phytoplasma_Onion_yellows:0.47843)
100:0.09099,(((((Listeria_monocytogenes_F2365:0.00063,
Listeria_monocytogenes_EGD:0.00144)90:0.00235,Listeria_innocua:0.00248)100:0.13517,
((Oceanobacillus_iheyensis:0.13838,Bacillus_halodurans:0.09280)
91:0.02676,(((Bacillus_cereus_ATCC_14579:0.00342,Bacillus_cereus_ATCC_10987:0.00123)
100:0.00573,Bacillus_anthracis:0.00331)100:0.08924,
Bacillus_subtilis:0.07876)96:0.01984)100:0.03907)69:0.02816,((Staphylococcus_aureus_MW2:0.00000,
(Staphylococcus_aureus_N315:0.00022,
Staphylococcus_aureus_Mu50:0.00022)61:0.00022)100:0.02479,
Staphylococcus_epidermidis:0.03246)100:0.17366)64:0.02828,
(((((((Streptococcus_agalactiae_III:0.00110,Streptococcus_agalactiae_V:0.00155)100:0.01637,
(Streptococcus_pyogenes_M1:0.00134,
(Streptococcus_pyogenes_MGAS8232:0.00045,(Streptococcus_pyogenes_MGAS315:0.00000,
Streptococcus_pyogenes_SSI-1:0.00022)100:0.00110)87:0.00066)
100:0.02250)100:0.01360,Streptococcus_mutans:0.04319)99:0.01920,
(Streptococcus_pneumoniae_R6:0.00119,Streptococcus_pneumoniae_TIGR4:0.00124)
100:0.03607)100:0.04983,Lactococcus_lactis:0.11214)100:0.08901,
Enterococcus_faecalis:0.07946)100:0.03958,(Lactobacillus_johnsonii:0.20999,
Lactobacillus_plantarum:0.14371)100:0.06763)100:0.08989)100:0.08905)92:0.09540)54:0.04315)
Bacteria:1.34959,(((((Thalassiosira_pseudonana:0.33483,
(Cryptosporidium_hominis:0.25048,Plasmodium_falciparum:0.28267)100:0.14359)42:0.03495,
(((Oryza_sativa:0.07623,Arabidopsis_thaliana:0.09366)
100:0.15770,Cyanidioschyzon_merolae:0.38319)96:0.08133,(Dictyostelium_discoideum:0.34685,
(((Eremothecium_gossypii:0.07298,Saccharomyces_cerevisiae:0.07619)
100:0.21170,Schizosaccharomyces_pombe:0.24665)100:0.15370,(((Anopheles_gambiae:0.10724,
Drosophila_melanogaster:0.10233)100:0.09870,
((Takifugu_rubripes:0.03142,Danio_rerio:0.05230)100:0.04335,(((Rattus_norvegicus:0.03107,
Mus_musculus:0.01651)91:0.00398,(Homo_sapiens:0.00957,
Pan_troglodytes:0.03864)100:0.01549)99:0.01629,Gallus_gallus:0.04596)100:0.01859)100:0.09688)
95:0.03693,(Caenorhabditis_elegans:0.01843,
Caenorhabditis_briggsae:0.01896)100:0.24324)100:0.09911)85:0.04004)41:0.02708)44:0.02636)
87:0.06455,Leishmania_major:0.45664)100:0.10129,
Giardia_lamblia:0.55482)100:0.57543,((Nanoarchaeum_equitans:0.81078,
(((Sulfolobus_tokodaii:0.17389,Sulfolobus_solfataricus:0.18962)100:0.33720,
Aeropyrum_pernix:0.43380)94:0.09462,Pyrobaculum_aerophilum:0.55514)100:0.12018)100:0.15444,
((Thermoplasma_volcanium:0.10412,Thermoplasma_acidophilum:0.09785)
100:0.66151,((((Methanobacterium_thermautotrophicum:0.36583,Methanopyrus_kandleri:0.35331)
99:0.07446,(Methanococcus_maripaludis:0.28592,
Methanococcus_jannaschii:0.13226)100:0.23828)100:0.06284,((Pyrococcus_horikoshii:0.02786,
Pyrococcus_abyssi:0.02179)100:0.02239,Pyrococcus_furiosus:0.02366)
100:0.36220)51:0.04469,(Archaeoglobus_fulgidus:0.34660,(Halobacterium_sp._NRC-1:0.61597,
(Methanosarcina_acetivorans:0.02602,Methanosarcina_mazei:0.03087)
100:0.30588)100:0.12801)100:0.10395)62:0.06815)99:0.11833)100:0.43325):0.88776);